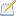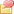R 包chipseeker安装失败

操作系统是Ubuntu24.04
R版本:4.3
需要在R下安装一些包:
chipseeker
clusterprofile
deseq2
尝试了安装方法都按照不上,
方法1:
在conda环境下安装:conda install bioconda::bioconductor-chipseeker
查看进程显示一直在下载包,

方法2:
按照官网的推荐的方式也安装不上
if (!requireNamespace("BiocManager", quietly=TRUE))
install.packages("BiocManager")
BiocManager::install("BiocUpgrade") ## you may need this
BiocManager::install("chipseeker")
Or the development version from github:
install.packages("devtools")
devtools::install_github("YuLab-SMU/ChIPseeker")
在 Ubuntu 24.04 上安装 R 包时遇到问题是常见的,特别是像 chipseeker、clusterProfiler 和 DESeq2 这样依赖于 Bioconductor 的包。你可以尝试以下步骤来解决问题:
方法 1:使用 R 的 Bioconductor 安装包
更新 R 包: 确保 R 的包管理器已经更新。打开 R 或 RStudio,然后运行以下命令:
R
复制代码
install.packages("BiocManager")
安装 Bioconductor 包: 使用 BiocManager 来安装所需的包。运行以下命令:
R
复制代码
BiocManager::install(c("ChIPseeker", "DESeq2", "clusterProfiler"))
解决依赖问题: 如果在安装过程中遇到任何依赖问题,可以使用以下命令来更新所有包:
R
复制代码
BiocManager::install()
方法 2:使用 conda 安装包
如果你在使用 conda 安装包时遇到问题,可以尝试以下步骤:
检查 conda 配置: 确保你已经添加了 Bioconda 和 conda-forge 通道。你可以通过以下命令来添加它们:
bash
复制代码
conda config --add channels bioconda
conda config --add channels conda-forge
创建新环境(可选): 为了避免依赖冲突,可以创建一个新的 conda 环境:
bash
复制代码
conda create -n myenv r-base=4.3
conda activate myenv
安装 R 包: 然后尝试再次安装包:
bash
复制代码
conda install bioconda::bioconductor-chipseeker bioconda::bioconductor-deseq2 bioconda::bioconductor-clusterprofiler
方法 3:手动安装
如果以上方法仍然无效,可以考虑手动下载包并安装:
下载包: 前往 Bioconductor 的官方网站,手动下载需要的包。
安装下载的包: 在 R 中使用以下命令安装:
R
复制代码
install.packages("path/to/downloaded/package.tar.gz", repos = NULL, type = "source")
方法 4:检查网络和依赖
网络问题:如果你在 conda 或 R 中下载包时速度很慢,检查网络连接,确保没有防火墙或代理阻止访问。
依赖问题:确认系统中安装了必要的依赖库,有时缺少某些系统库会导致包安装失败。可以通过以下命令安装常见的依赖:
bash
复制代码
sudo apt-get install libcurl4-openssl-dev libssl-dev libxml2-dev